Augur 分析
前言
IMPORTANT
Augur 是单细胞转录组学中用于细胞类型优先级排序的重要工具。它能够帮助研究者识别在特定扰动条件下最敏感的细胞类型,为理解细胞异质性背后的响应机制提供重要线索。
在单细胞研究中,我们不仅关注细胞的分类和标记基因,更希望深入了解不同条件下细胞的响应差异。Augur 通过机器学习算法评估不同细胞类型对扰动的敏感性,为识别关键细胞类型提供定量指标。
Augur 的核心功能
- 细胞类型优先级排序:基于机器学习算法评估不同细胞类型对扰动的敏感性
- 扰动响应可视化:将细胞类型优先级映射到降维图上,直观展示扰动响应的空间分布
- 定量评估指标:通过 AUC 值量化不同细胞类型对扰动的响应强度
- 跨条件比较:比较不同实验条件下细胞类型的扰动响应差异
本篇文档旨在为单细胞研究者提供一份详尽的 Augur 技术指南,内容涵盖其基本原理、在 SeekSoul Online 云平台上的操作方法、结果解读、实战案例及常见问题,帮助您快速掌握并应用该工具。
Augur 理论基础
核心原理
Augur 的核心思想是:通过机器学习算法评估不同细胞类型对扰动条件的响应敏感性,从而对细胞类型进行优先级排序。这一过程可以概括为以下几个主要步骤:
- 数据预处理:输入单细胞 RNA-seq 数据,选择要分析的细胞类型和扰动条件
- 特征选择:基于基因表达数据构建特征矩阵
- 机器学习建模:使用分类算法(如随机森林或逻辑回归)训练分类器,预测细胞的扰动状态
- 性能评估:通过受试者工作特征曲线下面积 (AUC) 评估分类器性能,AUC 值越高表示该细胞类型对扰动越敏感
- 优先级排序:根据 AUC 值对细胞类型进行排序,识别最敏感的细胞类型
关键算法详解
随机森林/逻辑回归 - 分类算法
- 原理:使用随机森林或逻辑回归算法基于基因表达特征预测细胞的扰动状态
- 输出:每个细胞类型的分类性能 (AUC 值),反映其对扰动的敏感性
AUC 评估 - 模型性能指标
- 原理:受试者工作特征曲线下面积 (Area Under the Curve) 用于评估分类器的性能
- 意义:AUC 值越高表示该细胞类型对扰动条件的响应越敏感
- 输出:每个细胞类型的 AUC 值,用于后续分析和可视化
核心分析指标
AUC (Area Under the Curve)
- 定义:受试者工作特征曲线下面积,衡量分类器性能的指标
- 计算:基于分类器对扰动状态的预测准确性
- 应用:量化不同细胞类型对扰动的敏感性,值越高表示越敏感
优先级排序 (Rank)
- 定义:基于 AUC 值对细胞类型进行的排序
- 计算:根据 AUC 值从高到低对细胞类型进行排序
- 应用:识别对扰动最敏感的细胞类型
云平台操作指南
在云平台上,Augur 分析流程被设计得直观易用。您无需编写代码,只需通过参数配置界面即可完成分析。
分析前的准备
IMPORTANT
Augur 分析的成功与否,很大程度上取决于输入数据的质量和扰动条件的合理性。在开始分析前,请务必确认:
- 数据已完成预处理:您的单细胞数据已经过标准的质控、降维、聚类和细胞类型注释。
- 扰动条件明确:明确要比较的扰动条件(如疾病 vs 健康、处理 vs 对照等)。
- 选择了合适的细胞亚群:Augur 分析应在具有生物学意义的细胞亚群中进行,如已注释的细胞类型。
参数详解
下表详细列出了云平台 Augur 分析模块的主要参数及其说明。
| 界面参数 | 说明 |
|---|---|
| 任务名称 | 本次分析的任务名称,需以英文字母开头。 |
| 分组因子 | 选择要分析的细胞类型或聚类对应的标签,例如 celltype。 |
| 细胞类型 | 选择要分析的具体细胞类型或聚类。 |
| 扰动因子 | 分组信息在 Seurat 对象的 meta.data 中的列名,用于定义扰动条件。 |
| 扰动对象 | 进行分析的扰动因子分组名称,至少选择两个。 |
| Downsample | 是否对大数据集进行随机下采样,默认为 False。 |
| Downsample_num | 每个细胞亚群下采样保留的细胞数目,默认为 1000。 |
| 备注 | 自定义备注信息。 |
重要注意事项
CAUTION
- 大数据集处理:当细胞总数较大时,若
Downsample参数设置为False,分析可能会因内存不足而失败。建议根据数据规模合理设置该参数。 - Metadata 规范:请确保 RDS 文件中的 metadata 列名和内容不包含中文或特殊字符(如
&),否则可能导致流程错误。 - 扰动因子选择:确保选择的扰动因子在 metadata 中存在且包含至少两个不同的分组。
操作流程
- 进入分析模块:在云平台导航至“高级分析”模块,选择“Augur”。
- 创建新任务:为您的分析任务命名,并选择要分析的样本或项目。
- 配置参数:根据上述指南,选择要分析的细胞类型、扰动因子和扰动对象等。
- 提交任务:确认参数无误后,点击“提交”按钮,等待分析完成。
- 下载与查看:分析结束后,在任务列表中下载并查看生成的分析报告和结果文件。
结果解读
Augur 的分析报告包含丰富的图表和数据文件,以下是对核心结果的详细解读。
可视化图表解读
AUC 降维图
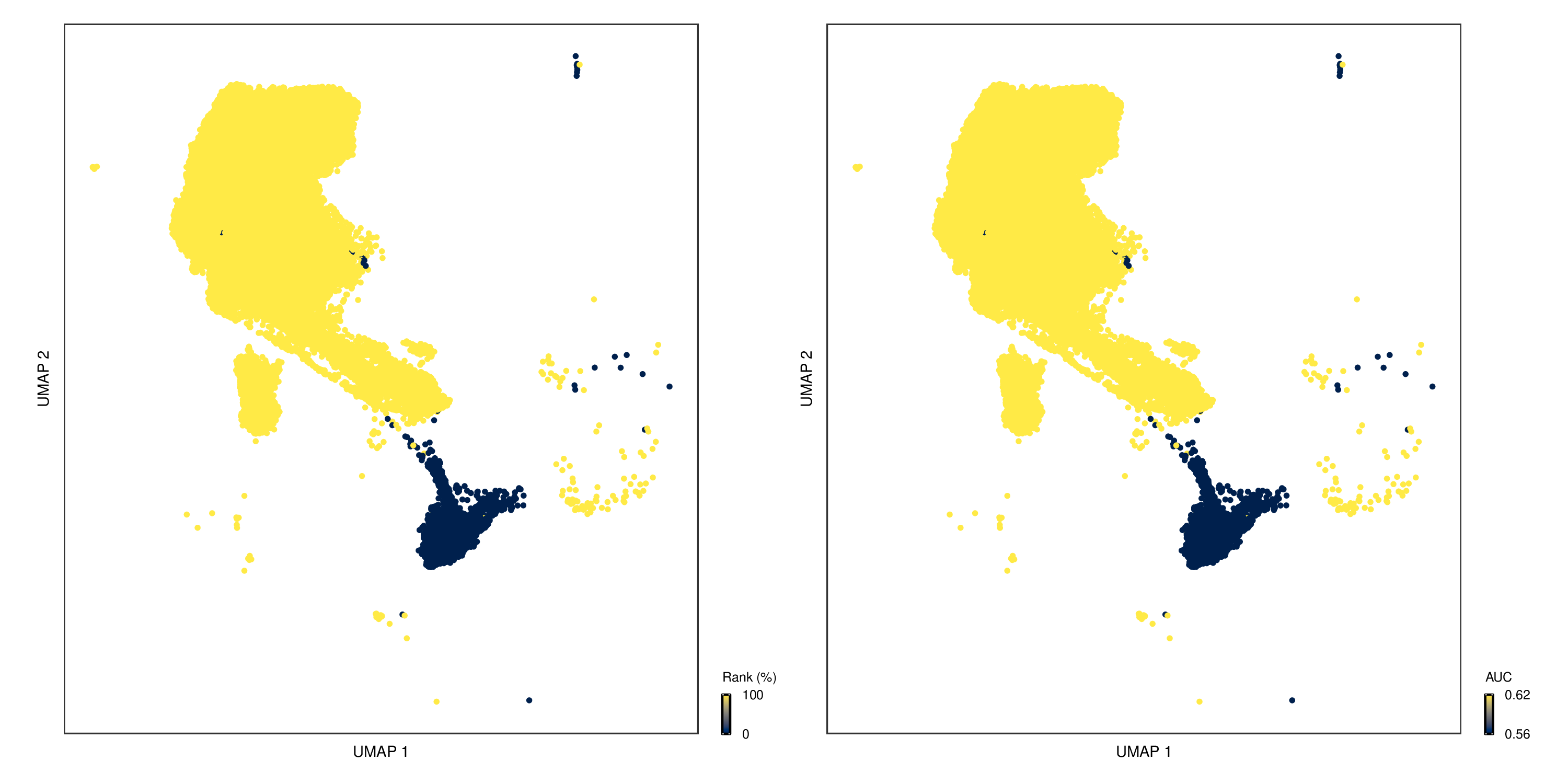
- 图表解读:在 UMAP/tSNE 图上展示不同细胞类型对扰动的响应敏感性。
- 颜色含义:黄色表示高 AUC 值(对扰动敏感),蓝色表示低 AUC 值(对扰动不敏感)。
- 左右图对比:
- 左图:基于 AUC 值的相对排名进行着色
- 右图:基于原始 AUC 值进行着色
AUC 棒棒糖图
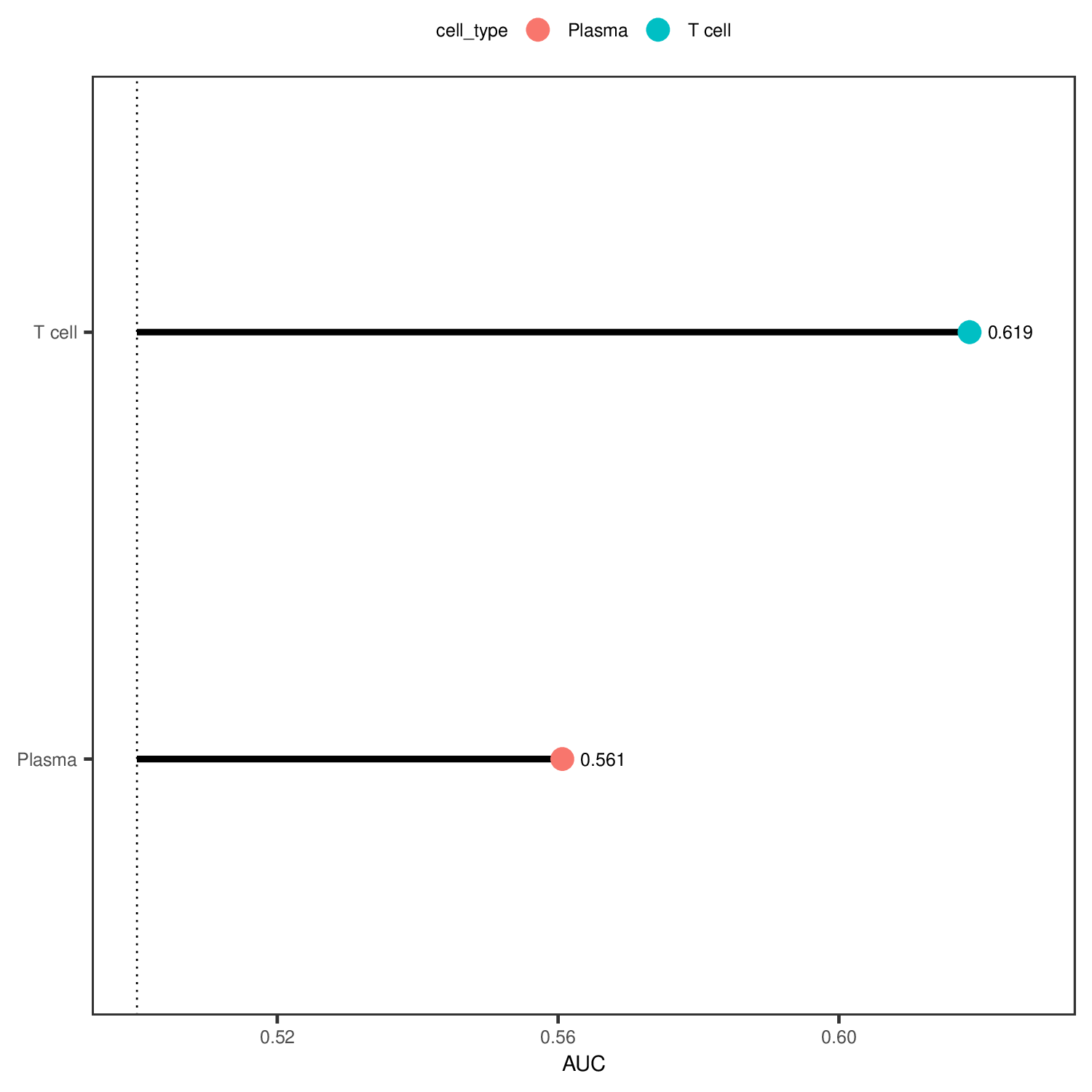
- 图表解读:展示各细胞类型的 AUC 值,用于比较不同细胞类型对扰动的敏感性。
- 横坐标:AUC 值
- 纵坐标:细胞类型
- 颜色含义:不同颜色代表不同细胞类型
AUC 箱线图
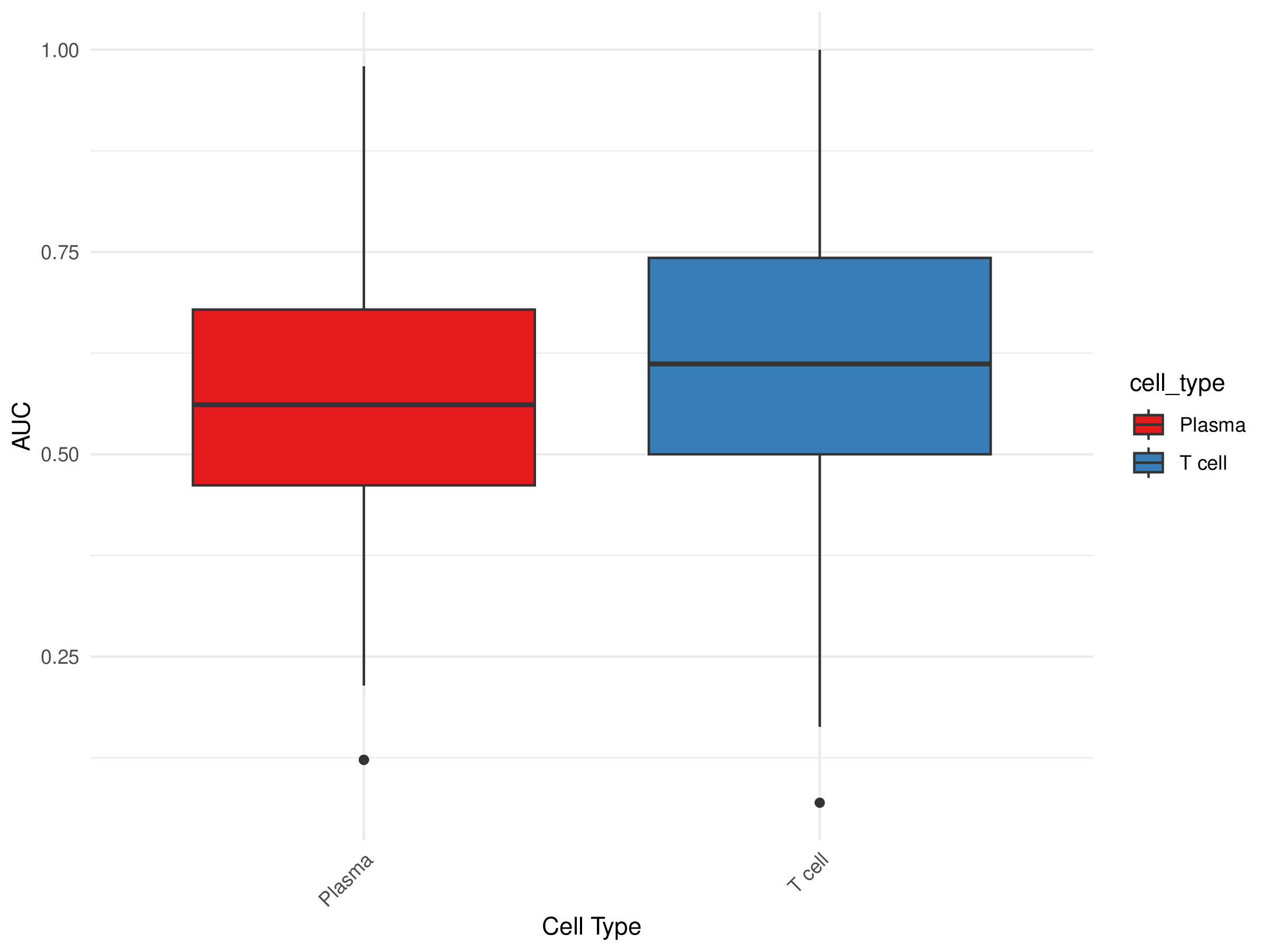
- 图表解读:展示各细胞类型 AUC 值的分布情况。
- 横坐标:细胞类型
- 纵坐标:AUC 值
- 颜色含义:不同颜色代表不同细胞类型
应用案例
案例一:脊髓损伤后行走功能恢复中的关键神经元识别
- 文献:Kathe C, Skinnider MA, Hutson TH, et al. Nature. 2022.
- 背景:目标是识别在脊髓损伤后行走功能恢复过程中起关键作用的神经元类型。
- 分析策略:使用 Augur 分析方法对 8 种实验条件下的 82,093 个细胞核转录组数据进行分析,对不同细胞类型进行优先级排序。
- 核心发现:
- 成功识别出表达 Vsx2 和 Hoxa10 的兴奋性脊髓中间神经元群 (SC_Vsx2::Hoxa10) 在硬膜外电刺激康复治疗中具有最高的 AUC 值。
- 通过功能验证实验证实,这些神经元在行走功能恢复过程中发挥着不可或缺的作用。
- 该研究展示了 Augur 在识别复杂生物过程中关键细胞类型方面的优势。
案例二:疾病状态下免疫细胞的扰动响应分析
- 背景:研究不同疾病状态下各类免疫细胞的响应敏感性差异。
- 分析策略:收集疾病组和健康对照组的单细胞 RNA-seq 数据,运行 Augur 分析比较不同免疫细胞亚群对疾病状态的响应敏感性。
- 核心发现:
- 识别出在疾病状态下响应最敏感的免疫细胞类型(如某些 T 细胞亚群或巨噬细胞亚群)。
- 这些高 AUC 值的细胞类型可能作为潜在的治疗靶点或生物标志物。
- 结合功能注释分析,揭示了关键细胞类型在疾病进展中的分子机制。
注意事项与最佳实践
TIP
避免过度解读:Augur 结果是基于转录组数据的计算推断,AUC 值的高低并不直接等同于生物学重要性。任何关键发现都需要后续的生物学实验来证实。
常见问题解答 (FAQ)
Q1: Augur 分析需要多长时间?
A: 分析时间取决于数据规模和计算资源配置。一般来说:
- 小数据集 (1,000-5,000 细胞):30 分钟 - 1 小时
- 中等数据集 (5,000-20,000 细胞):1-3 小时
- 大数据集 (> 20,000 细胞):3-8 小时或更长 建议根据数据规模合理设置 Downsample 参数以加快分析速度。
Q2: 如何选择合适的细胞类型进行分析?
A: 选择细胞类型时应考虑以下因素:
- 生物学意义:选择功能相关或在研究背景下感兴趣的细胞类型
- 数据质量:确保所选细胞类型具有足够的细胞数量 (建议 > 100 个细胞)
- 研究目的:根据具体研究问题选择感兴趣的细胞类型
Q3: AUC 值的意义是什么?
A:
- AUC (Area Under the Curve):受试者工作特征曲线下面积,用于评估分类器性能。在 Augur 中,AUC 值反映了细胞类型对扰动条件的响应敏感性。
- AUC 解释:AUC 值范围为 0.5-1.0,0.5 表示随机猜测水平,1.0 表示完美分类。通常 AUC > 0.7 被认为具有较好的分类性能。
Q4: 如何验证 Augur 分析结果的可靠性?
A: 可通过以下方式验证结果可靠性:
- 生物学验证:结合已知文献和数据库验证关键细胞类型的功能
- 实验验证:通过功能实验(如细胞功能抑制/激活实验)验证关键细胞类型的作用
- 交叉验证:使用不同的数据集或分析方法验证结果一致性
Q5: Augur 与其它细胞类型分析工具的区别?
A:
- Augur:专注于细胞类型对扰动条件的响应敏感性评估,通过机器学习算法进行优先级排序
- SCENIC:专注于基因调控网络推断和转录因子活性分析
- CytoTRACE:专注于细胞分化轨迹推断和干细胞识别
- Monocle:专注于拟时序分析和发育轨迹构建
Augur 官方仓库:https://github.com/neurorestore/Augur
参考资料
[1] SKINNIDER M A, SQUAIR J W, KATHE C, et al. Cell type prioritization in single-cell data[J]. Nat Biotechnol, 2021, 39(1): 30-34.
[2] KATHE C, SKINNIDER M A, HUTSON T H, et al. The neurons that restore walking after paralysis[J]. Nature, 2022, 611(7936): 540-547.
